BIATC受講生の声
今井 裕紀子
国立遺伝学研究所・小型魚類遺伝研究室 さきがけ専任研究員
(受講時:国立遺伝学研究所・小型魚類遺伝研究室 博士研究員)
私は,減数分裂期の相同組換えについて研究を行なっています.当時,私はゼブラフィッシュを新たな組換えモデルとすべく,研究を始めたところでした.この分野では,生殖細胞の核スプレッドを材料として,染色体に局在する様々な因子を免疫染色で解析することが欠かせません.こうした解析では,数百の細胞を観察し,マーカー因子の局在について定量的・統計的に議論することが必要です.画像解析のスループットを上げるために,Image Jのマクロについて勉強したいと思い,BIATC(生物画像データ解析トレーニングコース)を受講しました.マクロ習得を目指して参加しましたが,画像取得や画像処理についても自分の理解不足だった点に改めて気付かされました.本コースは単なるレクチャーではなく,「自分でできるようになること」に配慮されており,最終日には各受講者の様々な課題について講師の方々と議論する「クリニック」の時間も取られています.私の解析もクリニックに取り上げて頂き,テロメアと相同染色体の対合を定量するためのマクロについてご意見をもらいました.本コースでは画像解析を本質から理解できるので,学んだ内容をすぐに自分の解析に応用することができます.私も,講習で学んだことを土台としてマクロを作成し,減数分裂期の染色体構造変異体の表現型解析を客観的定量法で効率よく進めることができました [1, 2].マクロの導入によって,研究をスピードアップできたことは,ゼブラフィッシュの減数分裂研究を立ち上げる上で非常に大きな助けとなりました.これらの解析を通じて明らかになった「染色体末端指向的な組換え開始」というゼブラフィッシュの特徴は ,現在のさきがけ研究の出発点にもなっています [3].初心者はもちろんのこと,既に画像解析を行なっている人にとっても重要なTipsに満ちた講習です.是非,受講してワンランク上の解析を目指して下さい.
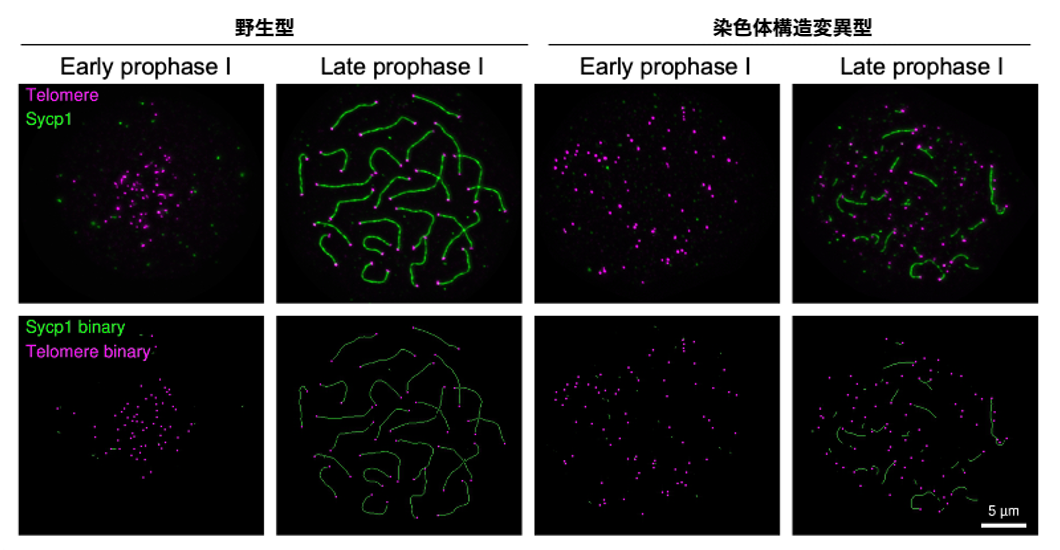
- Takemoto*, Imai* et al., (2020) Plos Genet DOI: 10.1371/journal.pgen.1008640 *co-first authors
- Imai et al. (2021) Front Cell Dev Biol DOI: 10.3389/fcell.2021.664377
- Imai et al. (2021) Front Cell Dev Biol (Review) DOI:10.3389/fcell.2021.757445